信息学院智能团队在国际重要学术期刊发表科研成果
校园网讯 近日,信息学院“复杂工业物流系统优化与控制”湖南省高校科技创新团队成员、“复杂工业物流系统智能控制与优化”湖南省重点实验室骨干潘理教授指导研究生王昊玥以第一作者在生物信息领域国际重要学术期刊《BMC Bioinformatics》发表了题为“A protein network refnement method based on module discovery and biological information”的学术论文,该论文针对蛋白质相互作用网络的细化方法,结合蛋白质在其中的模块化特征,提出了创新性方法,得到了同行专家的高度好评。
蛋白质是生物生命活动的主要承担者,其中关键蛋白质与生物细胞的生命活动密切相关。对关键蛋白质的识别有助于理解细胞生存的必需要求、发现药物靶点以及预防疾病。蛋白质通常无法独立完成某项功能,而是通过相互作用参与生命活动的各个方面,从而形成蛋白质相互作用网络(PIN)。基于PIN的关键蛋白质识别方法是当前生物信息学的研究热点。
从高通量生物实验中得到的PIN数据表现为较高的假阳性和假阴性,导致基于PIN的关键蛋白质识别方法很难获得理想的识别性能,因此如何通过网络细化方法过滤掉不可靠数据是提高网络质量的有效途径。研究表明,模块更有可能聚集关键蛋白质。但现有网络细化方法通常忽略PIN的模块特征。基于此,提出了一种基于模块发现和生物信息的蛋白质相互作用网络细化方法,尝试通过提高PIN质量来提升关键蛋白质识别性能。
方法的整体思想是,首先提取给定PIN中的最大连通子图,并通过Fast-unfolding算法将其划分为不同的模块;然后使用蛋白质的直系同源信息、亚细胞定位信息和模块在PIN中的拓扑信息来甄别关键模块;最后根据甄别的关键模块构建细化的蛋白质相互作用网络(CM-PIN)。
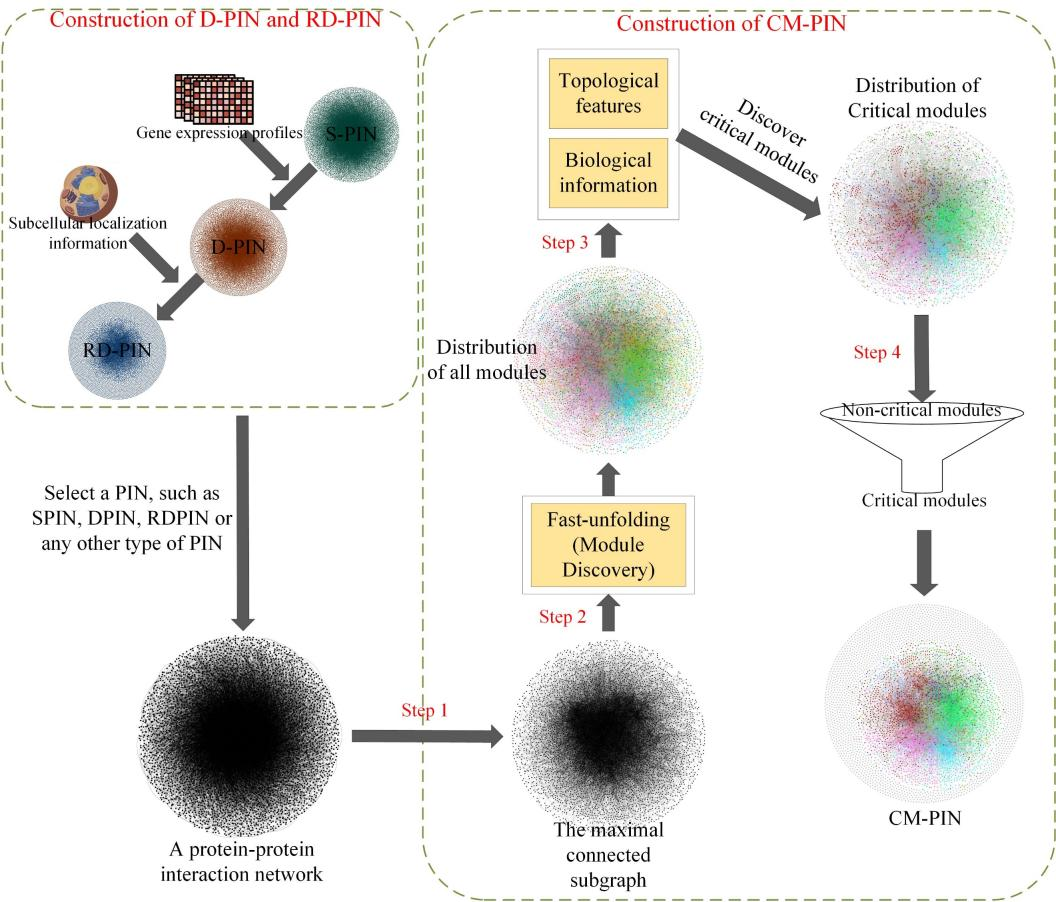
CM-PIN构建流程图
为了验证所提方法的有效性,选取了两个物种(酒酿酵母、人类),基于三种网络(静态蛋白质互作网络S-PIN、动态蛋白质互作网络D-PIN、细化的动态蛋白质互作网络RD-PIN),分别构建它们的CM-PIN,并通过12种节点排序方法,对比验证CM-PIN的关键蛋白质识别性能。实验结果表明,在Top100-Top600关键蛋白质的识别数、Jackknifing曲线、PRAUC值、敏感性、特异性、阳性预测值、阴性预测值、F测量值、Matthews相关系数和准确性等评估指标上,12种节点排序方法在CM-PIN中的表现都是最优的。

YDIP为例,各节点排序方法在个网络中的Jackknifing曲线
近年来,潘理教授在《IEEE Transactions on Systems, Man, and Cybernetics: Systems》《IEEE/ACM Transactions on Computational Biology and Bioinformatics》《BMC Bioinformatics》《电子学报》等期刊发表学术论文多篇,主持省级科研项目3项。
据悉,《BMC Bioinformatics》是生物信息学领域的重要学术期刊,中科院三区,CCF-C类,2023年影响因子为3.0。









 湘公网安备43060202000045号
湘公网安备43060202000045号